大家好,求教一个生物信息学的python编程。现需要用随机数按照染色体号和对应碱基数生成碱基序列。
染色体号与碱基数文件如下(txt文件)
chr01 1100
chr02 1400
chr03 1500
chr04 2200
chr05 1150
chr06 900
chr07 300
老师的代码如下:
import struct
import sys
import os
import random
args=sys.argv
def main(args):
chr_list = args[1]
fasta = args[2]
ATGC = ['A','T','G','C']
with open(chr_list,'r')as if_chr:
with open(fasta,'w')as of_fasta:
for line in if_chr:
chrome,length = line.strip('\n').split('\t')
length = int(length)
num = 0
of_fasta.write(">"+chrome+'\n')
print(chrome)
N=50
for i in range(1,length+N,N):
for k in range(0,N):
of_fasta.write(random.choice(ATGC))
num += 1
of_fasta.write('\n')
if name=="_main_":
main(args)
来一个运行代码图片: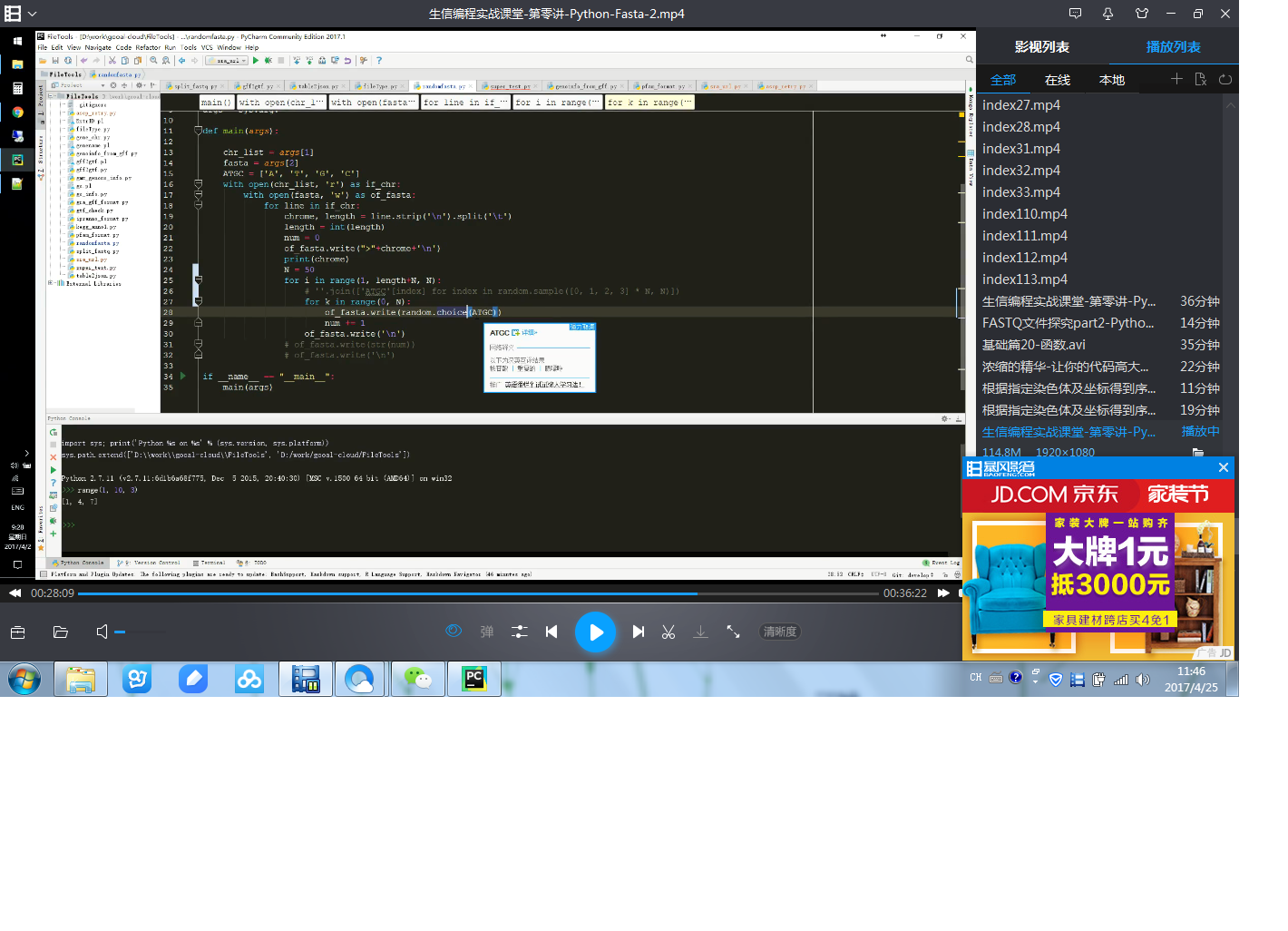
老师的python2运行前设置图片: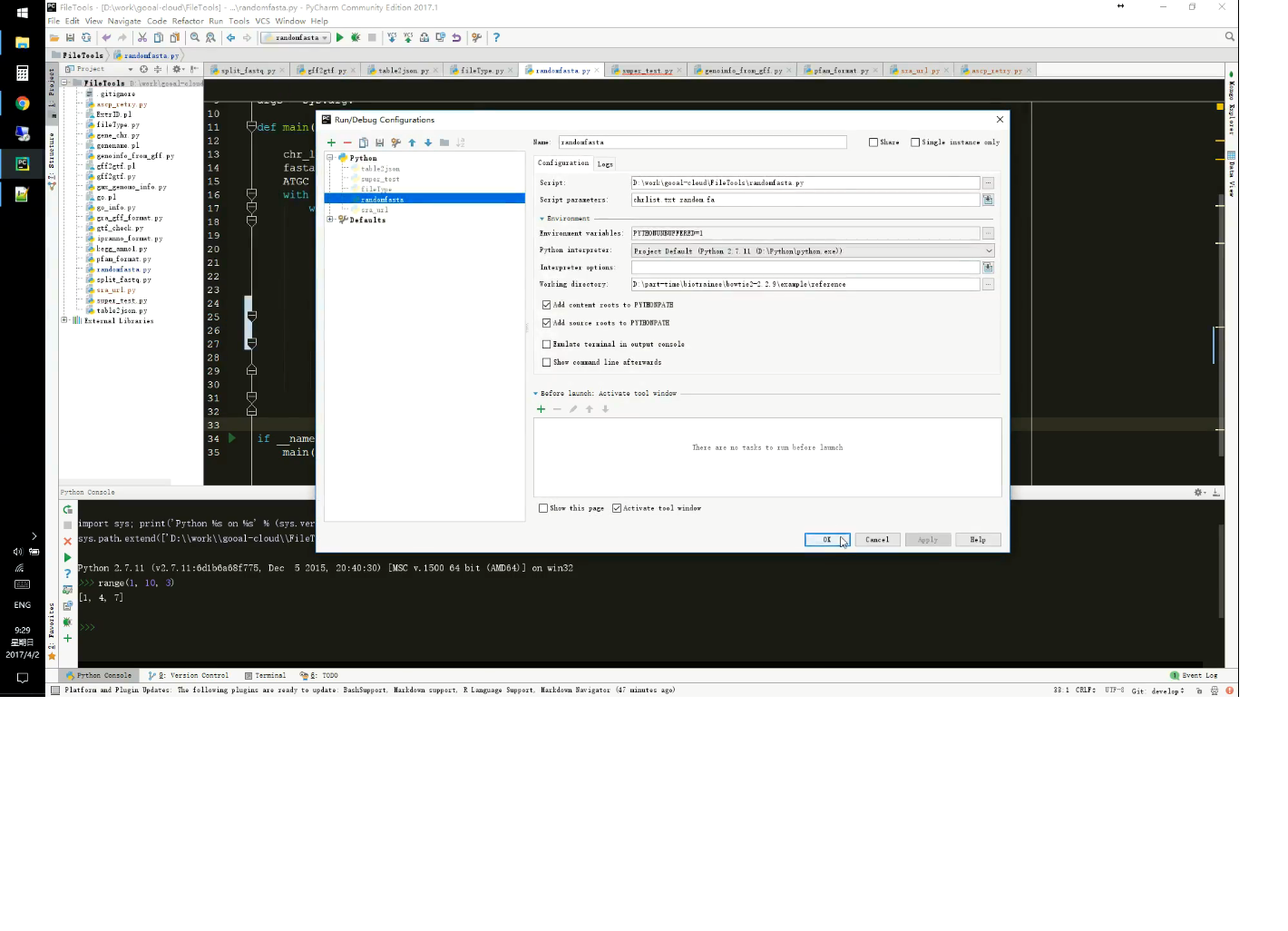
运行后可以生成结果: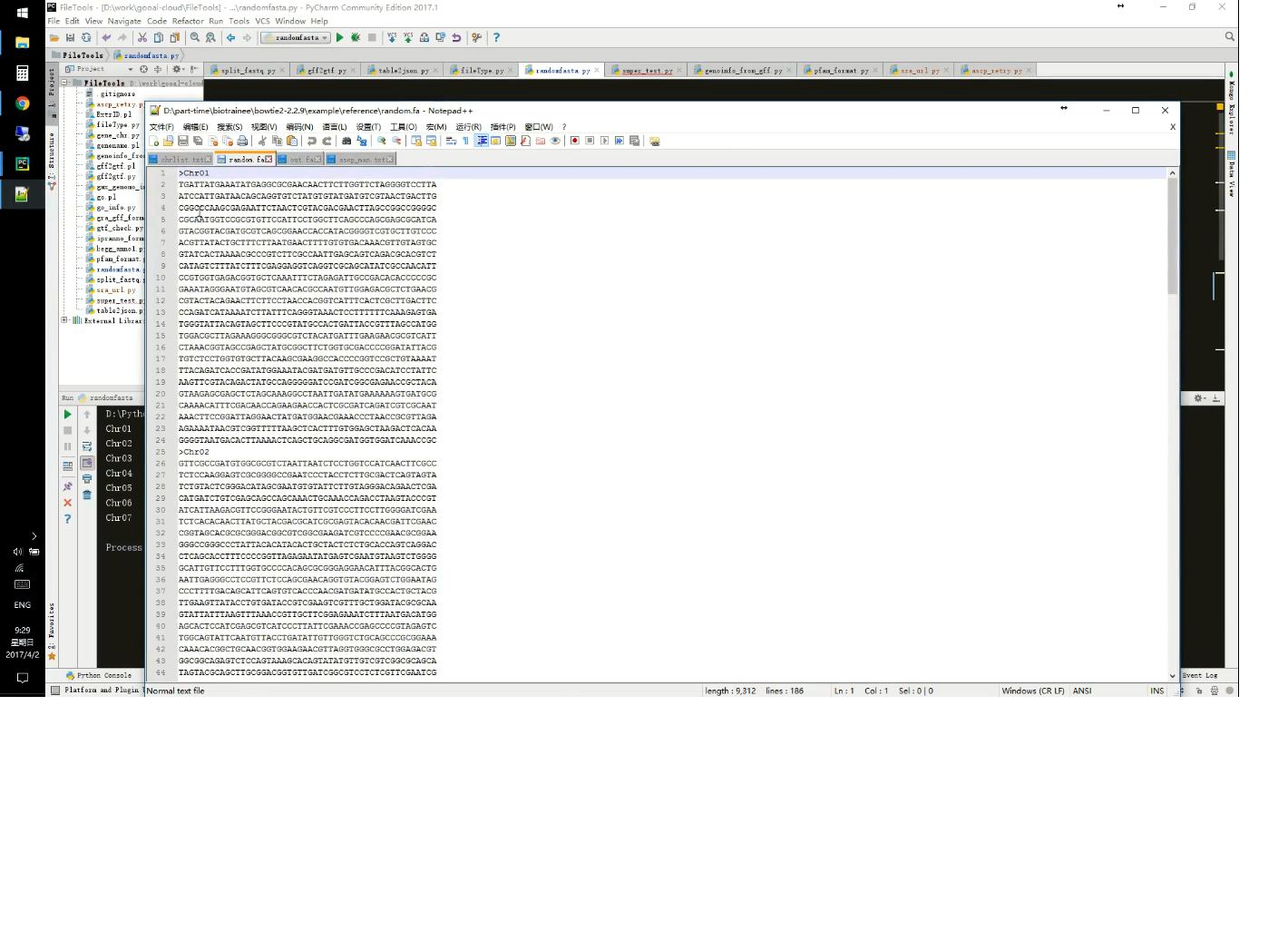
而我的Python3运行同样的代码结果是“NameError: name '_name_' is not defined
”如果把最后两行去掉,则显示“Process finished with exit code 0
”请专家指点迷津,到底是怎么回事。
为什么老师的Python2能产生一个TXT文件而我的python3却不能?
- 写回答
- 好问题 0 提建议
- 关注问题
- 邀请回答
-
3条回答 默认 最新
- 普通网友 2017-04-25 05:56关注
try this
if __name__=="__main__": main(args)本回答被题主选为最佳回答 , 对您是否有帮助呢?解决 无用评论 打赏 举报
