humann注释得到genefamilies文件,我已经转化为ko数据库的注释结果
如何根据数据库的注释结果,绘制了各样品在kegg各个功能层级上的相对丰度统计图,比如
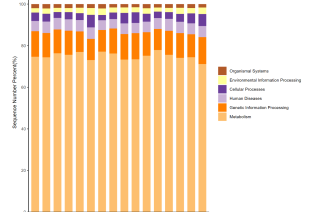
humann注释得到genefamilies文件,我已经转化为ko数据库的注释结果
如何根据数据库的注释结果,绘制了各样品在kegg各个功能层级上的相对丰度统计图,比如

哥哥要绘制 Humann2 的注释结果在 KEGG 数据库上的相对丰度统计图,您可以使用以下步骤:
genefamilies.tsv)。humann2_regroup_table 的工具,可以将基因家族重新映射到 KO。示例命令如下:humann2_regroup_table --input genefamilies.tsv --output ko_genefamilies.tsv --groups uniref90_ko
这将基于 KEGG 数据库中的 UniRef90 序列将 Gene Families 重新映射到 KO,并生成一个新的 KO 注释文件 ko_genefamilies.tsv。
需要注意的是,绘制 Humann2 结果的相对丰度统计图可能需要进行多次调整和优化,以满足您特定的研究问题和数据格式。但是,以上步骤应该可以帮助您入门并开始探索 Humann2 结果的可视化。