做甲基化上游,使用bismark进行reads比对。
现已完成建立基因组索引的步骤,预备进行正式比对。
输入
bismark --bowtie2 --path_to_bowtie2 /home/huanlab/anaconda3/envs/rna/bin --genome /home/huanlab/Met/bismark/experiencedata/log SRR517837C.fastq
看似成功执行该指令,但是BAM文件和report文件都是无内容的,如下图所示
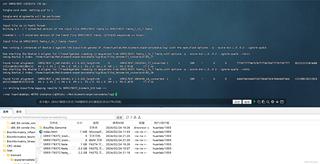
试了很多回,都一直这样。
bowtie2 版本2.51.和2.4.4都试过,目前bowtie2版本为2.4.4,bismark版本为0.24.2
请问大家有没有类似情况,又是如何解决的呢?感谢!

