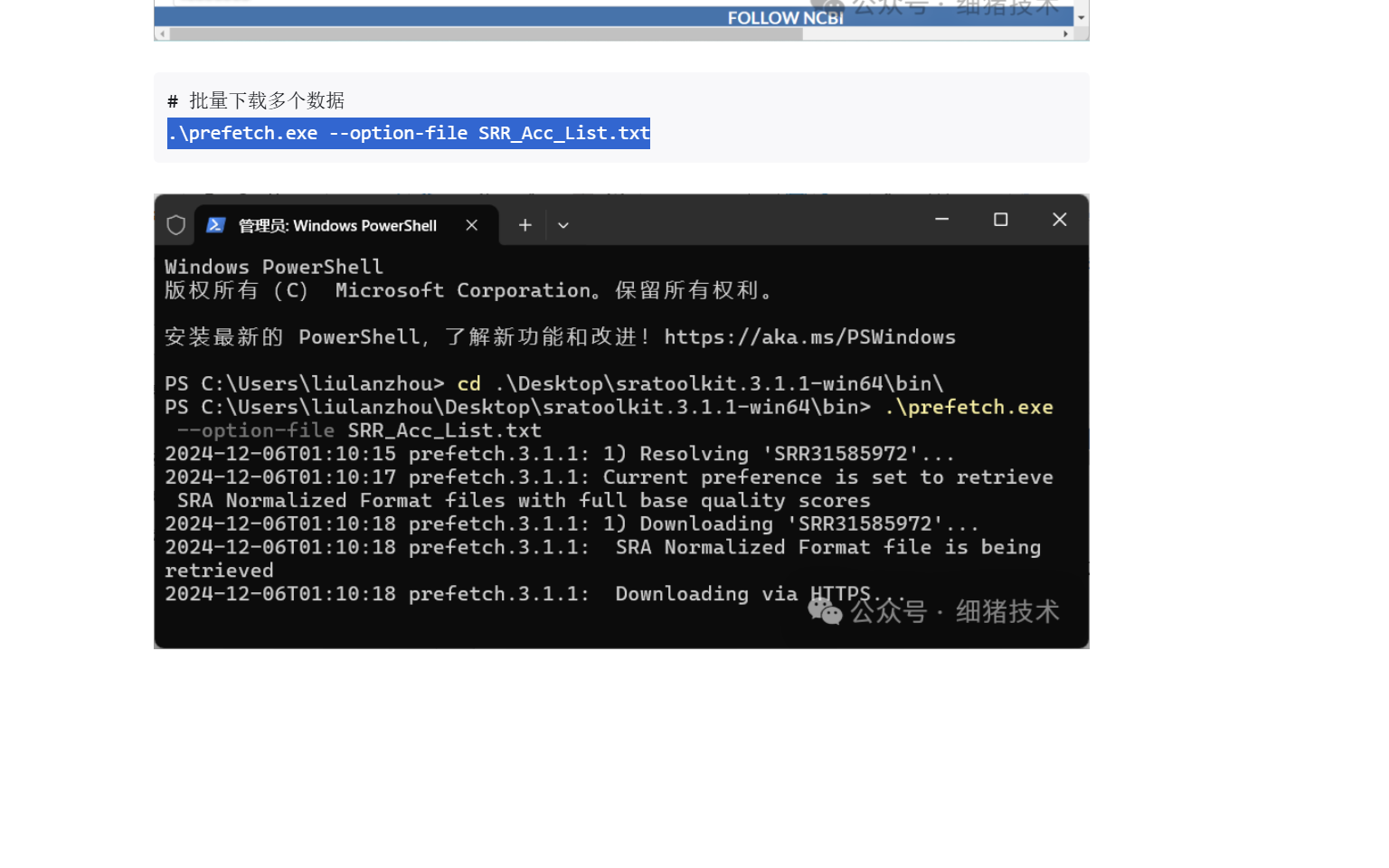
请问NCBI SRA数据下载 可批量下载SRR文件,下载完成后是否有SRR文件批量转fastq格式的方法

阿里嘎多学长整理AIGC生成,因移动端显示问题导致当前答案未能完全显示,请使用PC端查看更加详细的解答过程
你可以使用fastq-dump命令来批量转换SRR文件为fastq格式。fastq-dump是NCBI提供的命令行工具,可以从SRA数据库下载和转换SRA文件。
下面是一个示例命令:
fastq-dump --split-files --gzip --outdir output_dir SRR*.sra
其中:
--split-files 选项将SRA文件分割成多个fastq文件。--gzip 选项将fastq文件压缩为gzip格式。--outdir 选项指定输出目录。SRR*.sra 选项指定要转换的SRA文件名模式。你可以将这个命令保存到一个批处理文件中,然后在命令行中运行该文件来批量转换SRR文件。
如果你想使用Java来实现批量转换,可以使用SRA Toolkit的Java API。下面是一个示例代码:
import java.io.File;
import java.io.IOException;
import java.util.ArrayList;
import java.util.List;
import uk.ac.ebi.sra.sra-toolkit.ftp.FtpClient;
import uk.ac.ebi.sra.sra-toolkit.ftp.FtpFile;
import uk.ac.ebi.sra.sra-toolkit.ftp.FtpFileList;
import uk.ac.ebi.sra.sra-toolkit.ftp.FtpFileListFilter;
import uk.ac.ebi.sra.sra-toolkit.ftp.FtpFileListFilterFactory;
import uk.ac.ebi.sra.sra-toolkit.ftp.FtpFileListFilterFactory.FtpFileListFilterType;
import uk.ac.ebi.sra.sra-toolkit.ftp.FtpFileListFilterFactory.FtpFileListFilterType.FileListFilterType;
public class SRRToFastq {
public static void main(String[] args) throws IOException {
// 设置FTP客户端
FtpClient ftpClient = new FtpClient("ftp.ncbi.nlm.nih.gov", 21);
// 设置下载目录
String downloadDir = "/path/to/download/directory";
// 设置输出目录
String outputDir = "/path/to/output/directory";
// 获取SRR文件列表
FtpFileList ftpFileList = ftpClient.listFiles(downloadDir, new FtpFileListFilterFactory().createFilter(FtpFileListFilterType.REGEX, "SRR.*\\.sra"));
List<File> srrFiles = new ArrayList<>();
for (FtpFile ftpFile : ftpFileList) {
srrFiles.add(new File(downloadDir, ftpFile.getName()));
}
// 批量转换SRR文件为fastq格式
for (File srrFile : srrFiles) {
String fastqFile = outputDir + "/" + srrFile.getName().replace(".sra", ".fastq.gz");
FtpClient ftpClient2 = new FtpClient("ftp.ncbi.nlm.nih.gov", 21);
ftpClient2.downloadFile(srrFile.getAbsolutePath(), fastqFile);
}
}
}
这个示例代码使用SRA Toolkit的Java API来连接FTP服务器,获取SRR文件列表,然后批量下载和转换SRR文件为fastq格式。
请注意,这个示例代码只是一个简单的示例,实际实现中可能需要根据你的需求进行修改和调整。