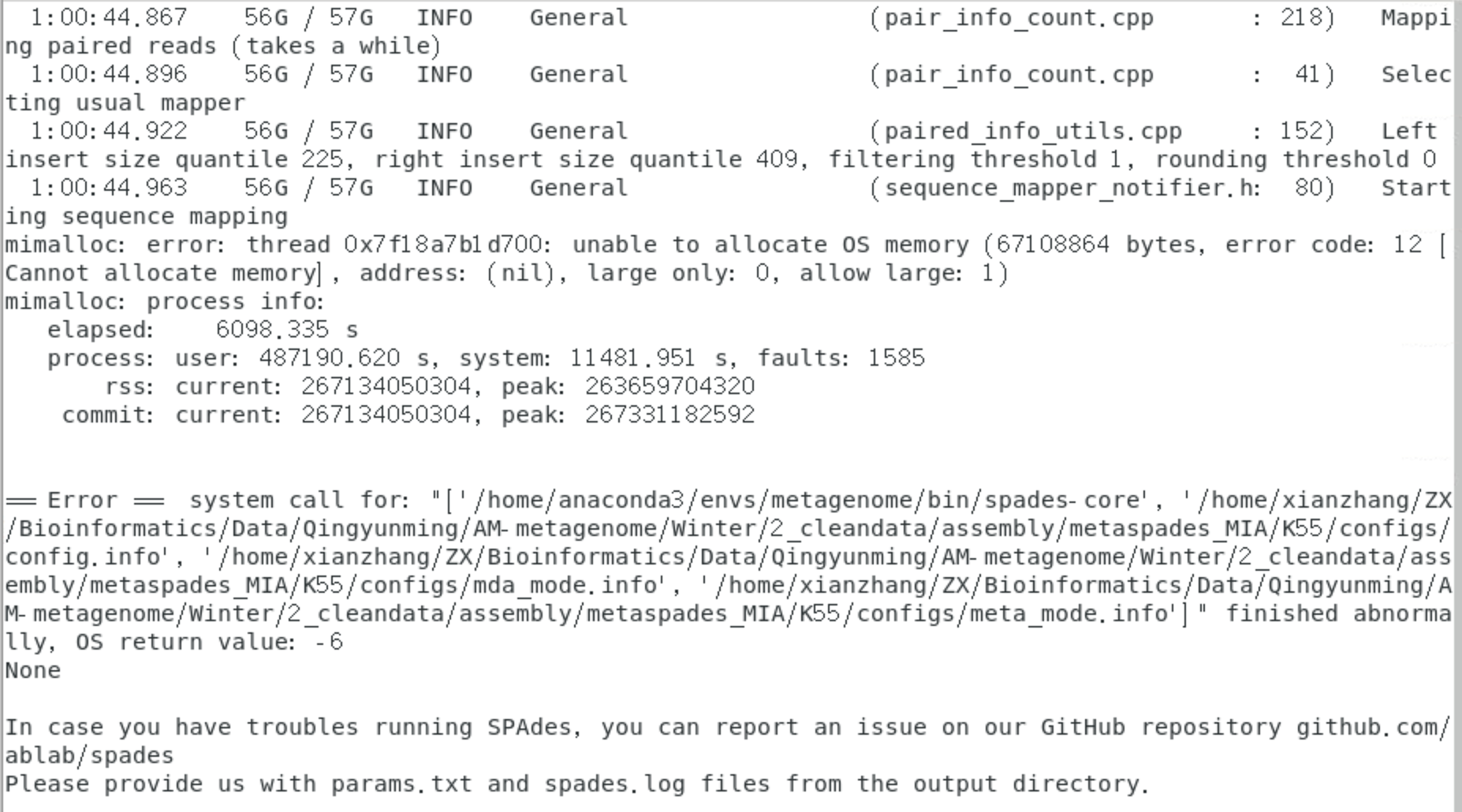
== Error == system call for: "['/home/anaconda3/envs/metagenome/bin/spades-core', '/home/xianzhang/ZX/Bioinformatics/Data/Qingyunming/AM-metagenome/Winter/2_cleandata/assembly/metaspades_MIA/K55/configs/config.info', '/home/xianzhang/ZX/Bioinformatics/Data/Qingyunming/AM-metagenome/Winter/2_cleandata/assembly/metaspades_MIA/K55/configs/mda_mode.info', '/home/xianzhang/ZX/Bioinformatics/Data/Qingyunming/AM-metagenome/Winter/2_cleandata/assembly/metaspades_MIA/K55/configs/meta_mode.info']" finished abnormally, OS return value: -9
有朋友做过metaspades的宏基因组组装吗,我7个样本只剩这个样本总是跑不出来,比这个样本数据大一倍的样本都跑出来了,这个样本同样的参数或者改了参数都跑不出来,要么是== Error == system call for:“...“finished abnormally, OS return value: -9要么是finished abnormally, OS return value: -11或者-6