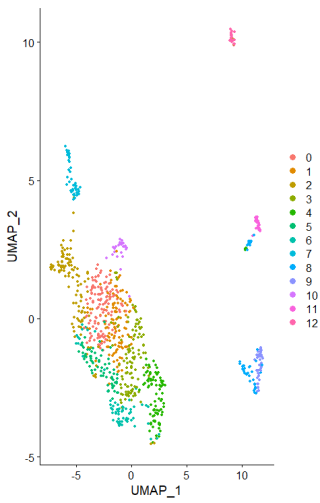
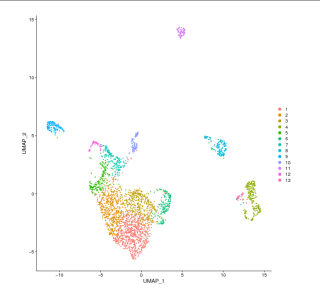
#细胞进行聚类
在做单细胞seurat分析的时候进行细胞聚类如何设置让细胞分类从1开始,而不是从0开始呢
#细胞进行聚类
pbmc <- FindNeighbors(object = pbmc, dims = 1:10)
pbmc <- FindClusters(object = pbmc, resolution = 1.3)
# Look at cluster IDs of the first 5 cells
head(x = Idents(object = pbmc), 5)
p1 <- DimPlot(pbmc, reduction = "umap")
p1