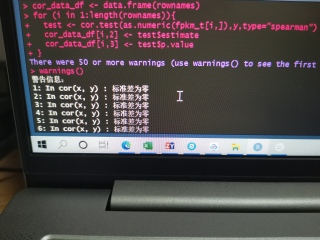
R语言用fpkm文件做基因相关性分析警告,提示标准差为0,哭了,但是还是有相关性结果表出来,后面相对两个基因相关性作散点图,又报错,该怎么办
R语言用fpkm文件做基因相关性分析警告,提示标准差为0,哭了,但是还是有相关性结果表出来,后面相对两个基因相关性作散点图,又报错,该怎么办



关于第一个问题,出现警告信息与fpkm_t[i,]及y两个变量有关,其中一个变量可能值都相同,或者原数据就存在问题不适宜做相关分析,输出两个变量看看,并检查前面数据处理是否有问题。对于这种报错可以看看这里的原因解释,相应地对x,y参数做查验和调整。
https://statisticsglobe.com/r-warning-message-cor-standard-deviation-is-zero
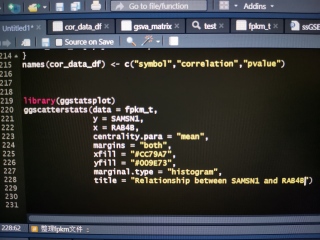
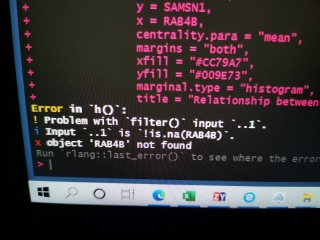
第二个问题,查看一下传入函数的参数x,y是否顺序颠倒了或者看看数据行列的顺序是否正确。
如有帮助,请点采纳。