R语言,使用单因素cox检验发现不符合ph假设,于是对其进行时间分层,分层后想再次进行ph假设检验,报错。
这是代码
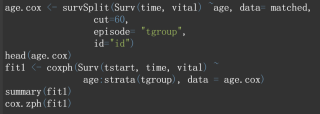
这是返回的结果
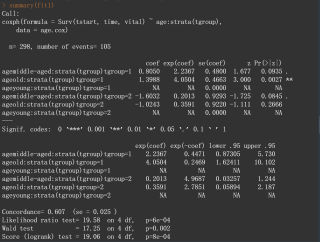
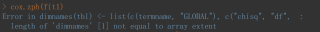
大家帮忙看下是什么问题,怎么修改,感谢!
R语言,使用单因素cox检验发现不符合ph假设,于是对其进行时间分层,分层后想再次进行ph假设检验,报错。
这是代码

这是返回的结果


该回答引用于gpt与OKX安生共同编写:
根据您提供的信息,可能是因为使用了cox.zph()函数时,有些分层的时间段内没有事件发生,导致出现了分母为0的情况,从而导致了错误。
解决这个问题的方法是在每个时间段中加入至少一个事件,可以通过在数据中添加一些虚拟事件来实现。具体步骤如下:
cox.zph()函数的假设检验。以下是示例代码:
# 加载survival包
library(survival)
# 读取数据
data <- read.csv("data.csv")
# 复制数据用于添加虚拟事件
data_with_virtual_events <- data
# 时间分层
cutpoints <- c(12, 24, Inf)
data_with_virtual_events$time_group <- cut(data_with_virtual_events$time, breaks = cutpoints, labels = c("12", "24"))
# 在每个时间段末尾添加一个虚拟事件
virtual_events <- data.frame(time = c(12.5, 24.5), status = rep(0, 2))
data_with_virtual_events <- rbind(data_with_virtual_events, virtual_events)
# 单因素Cox回归
fit <- coxph(Surv(time, status) ~ factor(group), data = data_with_virtual_events)
# 假设检验
test_result <- cox.zph(fit)
在上面的代码中,需要替换"data.csv"为实际的数据文件名,并根据实际的时间分层方式调整cutpoints参数。另外,由于添加了虚拟事件,最终得到的结果中可能会存在贡献为0的时间段,需要根据实际需求进行后处理。
数据中存在缺失值。如果在进行Cox回归之前没有对数据进行处理,那么可能会出现缺失值而导致对照组为NA。可以通过删除包含缺失值的行或使用均值、中位数等方法填充缺失值来解决。
在进行因子变量转换时,可能遗漏了某些水平,导致某个水平被当做对照组,但是该水平确实不存在于数据中。此时需要检查因子变量转换的代码是否正确。
在进行因子变量转换时,可能没有指定哪个水平作为对照组,导致R默认将字母排序最小的水平作为对照组。如果希望指定其他水平作为对照组,则可以使用relevel函数来更改对照组。
下面是一个使用relevel函数指定因子变量的某个水平作为对照组的例子:
# 假设有一个名为"factor_var"的因子变量,其中包含a、b、c三个水平
# 将"b"设为对照组
data$factor_var <- relevel(data$factor_var, ref = "b")
这样,在进行Cox回归分析时,R就会将"b"作为对照组,而不是默认的字母排序最小的水平。
希望以上信息能对你解决问题有所帮助。