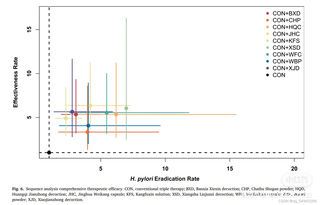
网状meta分析,有代码包吗,跟gemtc包有关吗,有没有推荐代码店铺
 关注
关注引自免费微信小程序:皆我百晓生
根据您提供的图示信息,这似乎是一个用于展示不同疗法对幽门螺杆菌根除率效果的网状meta分析图。这种类型的分析和可视化通常在R语言中可以通过networkmeta、netmeta或者gemtc等包实现。特别是gemtc包是专门用于贝叶斯网状Meta分析(Bayesian Network Meta-Analysis, NMA)的工具。
要进行网状meta分析并生成类似图表,可以使用gemtc包结合ggplot2或其他绘图包来绘制。以下是一个基本的示例代码流程:
# 安装所需的R包
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("gemtc")
# 加载相关包
library(gemtc)
library(netmeta)
# 假设你已经有了一个包含治疗效果数据的数据框df,并且格式化为适合gemtc包输入的形式
# df <- ... # 这里应填充您的数据
# 构建网状Meta分析模型
mtc.model <- mtc.network(data.df = df)
# 执行贝叶斯分析
res <- mtc.run(mtc.model)
# 结果可视化
plot(res, show.values = TRUE)
# 或者更灵活地使用ggplot2进行自定义绘图
library(ggplot2)
ggplot(res$summary, aes(x = treatment, y = effect, ymin = lower, ymax = upper)) +
geom_pointrange() +
coord_flip() +
labs(x = "Therapy", y = "H. pylori Eradication Rate") +
theme_bw()
请注意,上述代码仅作为基本示例,实际应用时需要根据您的具体数据集调整数据处理和分析步骤。同时,您可能还需要从原始研究中提取并整理相应的效应量(如OR、RR等)、置信区间以及相关的变异量信息,才能完成整个分析过程。
至于推荐的代码店铺或资源,通常学术论坛、GitHub上的开源项目以及Stack Overflow等网站会有很多与R语言及特定包相关的实例代码和教程,您可以自行搜索查阅以获取更多帮助。