出现这个错误应该怎么解决?gt11是large matrix形式,as.data.frame(gt11)后还是这个错误
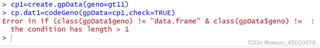
> cp1=create.gpData(geno=gt11)
> cp.dat1=codeGeno(gpData=cp1,check=TRUE)
Error in if (class(gpData$geno) != "data.frame" & class(gpData$geno) != :
the condition has length > 1
出现这个错误应该怎么解决?gt11是large matrix形式,as.data.frame(gt11)后还是这个错误

> cp1=create.gpData(geno=gt11)
> cp.dat1=codeGeno(gpData=cp1,check=TRUE)
Error in if (class(gpData$geno) != "data.frame" & class(gpData$geno) != :
the condition has length > 1
该回答引用自GPT-3.5,由博主GISer Liu编写:
根据报错信息 "the condition has length > 1",这个错误表明条件的长度大于1。在代码中,出现这个错误的地方是在检查输入参数gpData 的 geno 属性是否为数据框架类型时。然而,出现了长度大于1的情况,可能是因为 gpData
的 geno 属性返回了一个长度大于1的结果,这与预期的数据框架类型不符。
解决这个问题的思路是确保 gpData 的 geno 属性返回的数据是一个单一的数据框架,而不是长度大于1的对象。
以下是一种可能的解决方案:
RCopy code
# 首先检查 gpData$geno 的类型
class(gpData$geno)
# 如果是长度大于1的对象,则将其转换为数据框架
if (length(gpData$geno) > 1) {
# 转换为数据框架
gpData$geno <- as.data.frame(gpData$geno)
}
# 然后再次检查类型
class(gpData$geno)
# 如果是数据框架类型,则继续执行 codeGeno 函数
cp.dat1 <- codeGeno(gpData = cp1, check = TRUE)
这段代码首先检查 gpData$geno
的类型,如果发现长度大于1,则将其转换为数据框架类型。然后再次检查类型,确保已经转换为正确的类型后,再继续执行 codeGeno 函数。
通过这种方式,应该能够解决报错问题。
如果该回答解决了您的问题,请采纳!如果没有,请参考以下方案进行修订
