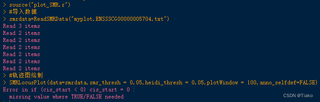
有的基因能画出轨迹图和相关图,有的基因就会报错,如上图,这是什么原因呢?能画出图的数据也有缺失值

以下内容由CHATGPT及阿里嘎多学长共同生成、有用望采纳:
错误信息 "Error in if (cis_start < 0) cis_start = 0 : missing value where TRUE/FALSE needed" 表示在执行 R 语言代码时,if 语句中的条件判断遇到了一个缺失值(NA),导致无法判断为 TRUE 或 FALSE。这通常发生在数据预处理或数据读取阶段,当数据集中存在缺失值时,需要在进行条件判断之前对其进行处理。
针对你的问题,以下是一些可能的解决方案:
检查数据:首先检查 smrdata 数据集,确认是否存在缺失值(NA)。可以使用以下代码来检查:
is.na(smrdata)
处理缺失值:如果发现缺失值,需要决定如何处理它们。常见的处理方法包括:
修改条件判断:在 if 语句中添加对缺失值的检查,例如:
if (!is.na(cis_start) && cis_start < 0) {
cis_start = 0
}
这样可以确保只有当 cis_start 不是缺失值且小于 0 时,才会将其设置为 0。
检查数据输入:确保读取的数据文件 "myplot.ENssSCG00000005704.txt" 是正确的,并且格式符合 ReadShRData 函数的要求。如果文件格式不正确或数据损坏,也可能导致错误。
查看函数文档:查看 SMRLocusPlot 和 ReadShRData 函数的文档,了解它们对输入数据的具体要求和处理方式。
调试代码:逐步执行代码,检查每一步的输出,找出导致错误的具体位置。
寻求帮助:如果问题依然无法解决,可以寻求社区的帮助,例如在 Stack Overflow 或 R 语言相关的论坛发帖询问。
关于参考资料,以下是一些可能有用的链接:
请注意,由于我无法访问外部链接,上述链接需要你自行访问。希望这些建议能帮助你解决问题。如果你需要进一步的帮助,请提供更多的代码和数据信息。