使用PredictProtein预测蛋白质的二级结构,结果如图,该怎么看?
 关注
关注引自免费微信小程序:皆我百晓生
在理解PredictProtein的结果时,你需要分析几个关键部分:二级结构、 solvent accessibility(溶剂可及性)、拓扑信息、以及一些特定的功能区域和特性。下面是对图1和图2内容的详细解释:
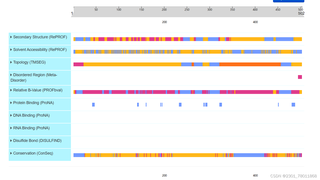
图1:
Secondary Structure (RePROF):这部分显示了预测的二级结构,包括α螺旋(H),β折叠(E),无规卷曲(C)以及不确定的区域(.)。这些字母表示不同的二级结构类型。
Solvent Accessibility (RePROF):这个区域给出了氨基酸残基的溶剂可及性,通常以百分比表示。高值(接近100%)代表残基暴露于溶剂,低值(接近0%)则表示残基埋藏在蛋白内部。
Topology (TMSEG):这部分提供了关于膜蛋白拓扑的信息,例如跨膜段的位置。如果你的蛋白质是膜蛋白,这里会显示跨膜段的开始和结束位置。
Disordered Region (Meta-Disorder):这是对蛋白质中可能的无序区域的预测,无序区域不具有稳定的二级结构。
Relative B-Value (PROFbval):B值是一个反映原子运动性的参数,与结构稳定性和灵活性有关。较高的B值对应较高的动态性,较低的B值对应较高的稳定性。
Protein Binding (ProNA), DNA Binding (ProNA), RNA Binding (ProNA):这三部分分别显示了预测的蛋白质相互作用、DNA结合位点和RNA结合位点的可能性。
Disulfide Bond (DISULFIND):这里显示了预测的二硫键位置,二硫键是由两个半胱氨酸残基形成的化学键,对于稳定蛋白质结构很重要。
Conservation (ConSeq):这一项展示了序列保守程度,高值意味着该区域在同源蛋白中的氨基酸组成相似度很高,可能有重要的功能。
图2:
Zoom-Start: 1, End: 502: 这部分告诉你当前视图覆盖的氨基酸范围是从第1到第502个残基。
Export to image: 这个选项允许你将当前的图形导出为图片文件。
为了更直观地理解这些信息,你可以尝试使用像Weblogo(https://weblogo.berkeley.edu/logo.cgi)这样的工具来创建一个可视化的蛋白质结构图,输入PredictProtein提供的二级结构预测结果。同时,你也可以使用专门的蛋白质结构可视化软件,比如PyMOL(https://pymol.org/)或 ChimeraX(https://www.cgl.ucsf.edu/chimerax/)来查看整个蛋白质模型,并叠加这些预测信息。
如果你想要自己编写Python脚本来解析PredictProtein的输出并绘制简单的图表,可以参考以下代码片段(注意:这个例子仅用于展示如何读取数据,实际绘图可能需要更复杂的库,如matplotlib):
# 假设你的数据存储在一个名为'predictions.txt'的文本文件中
with open('predictions.txt', 'r') as f:
lines = f.readlines()
# 提取二级结构部分
secondary_structure_lines = lines[6:-2] # 前面几行是标题,最后一行是空行
secondary_structure = ''.join(line.strip() for line in secondary_structure_lines)
print("二级结构预测:", secondary_structure)
要完全解析所有其他信息并进行可视化,你可能需要更深入地了解每个模块的输出格式,并可能需要写更复杂的代码来处理这些数据。建议参考 PredictProtein 的官方文档或相关的研究论文来获取更准确的解析方法。