哑铃图显示某i个基因在不同样本不同细胞类型中的表达量,但是样本排序是按照首字母排序,不是按照生物学排序。
VlnPlot(data, features = c("DUSP2"), split.by ="orig.ident" )
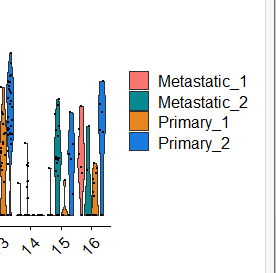
哑铃图显示某i个基因在不同样本不同细胞类型中的表达量,但是样本排序是按照首字母排序,不是按照生物学排序。
VlnPlot(data, features = c("DUSP2"), split.by ="orig.ident" )

晚上好🌙🌙🌙
本答案参考通义千问
在使用 VlnPlot 绘制哑铃图(或小提琴图)时,如果发现样本排序是按照首字母顺序排列,而不是根据生物学意义(如实验批次、处理条件等)进行排序,这可能会影响对数据的解读。为了解决这个问题,你可以通过 调整 split.by 的因子顺序 来实现自定义样本排序。
你目前使用的代码如下:
VlnPlot(data, features = c("DUSP2"), split.by = "orig.ident")
其中 orig.ident 是样本标识符(例如:SampleA, SampleB, SampleC),默认情况下,R 会将它们按字母顺序排序(即 SampleA, SampleB, SampleC)。但你希望按照 生物学逻辑 排序,比如 Control, Treatment1, Treatment2 等。
orig.ident 转换为因子,并指定正确的顺序# 假设你的样本名称是:Control, Treatment1, Treatment2
data$orig.ident <- factor(data$orig.ident, levels = c("Control", "Treatment1", "Treatment2"))
注意: 你需要根据实际的样本名替换上面的
levels参数。
VlnPlot(data, features = c("DUSP2"), split.by = "orig.ident")
这样,样本将会按照你指定的顺序展示。
str(data$orig.ident)
确保输出显示的是你期望的顺序,例如:
Factor w/ 3 levels "Control","Treatment1","Treatment2": ...
# 假设你的数据对象是 data,且 orig.ident 是样本列
# 步骤1:设置样本顺序
data$orig.ident <- factor(data$orig.ident, levels = c("Control", "Treatment1", "Treatment2"))
# 步骤2:绘制哑铃图
VlnPlot(data, features = c("DUSP2"), split.by = "orig.ident")
| 步骤 | 操作 | 说明 |
|------|------|------|
| 1 | 将 orig.ident 转换为因子 | 指定样本的排序顺序 |
| 2 | 使用 VlnPlot 绘图 | 根据新因子顺序展示样本 |
| 3 | 可选:检查因子顺序 | 确保排序符合预期 |
如果你能提供具体的样本名称,我可以进一步帮你定制 levels 参数。