为什么我的inferCNV跑出来分层聚类树里永远只有observed这一种,我的annotation里面明明是T/NK作为参照以及0-9群上皮细胞,想判断0-9群的恶性成都,但是做出来下面observed里面没有0-9群的分群显示
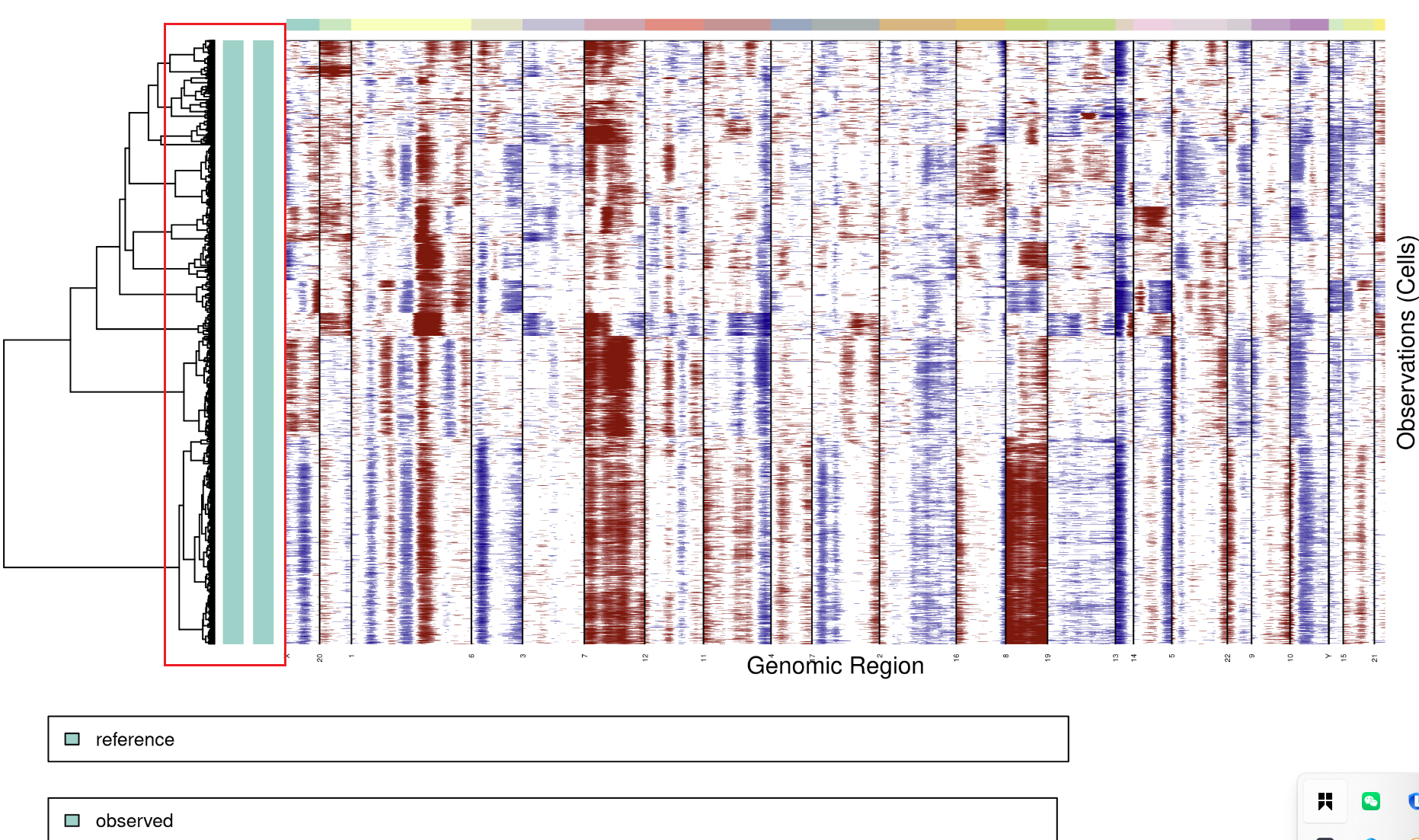
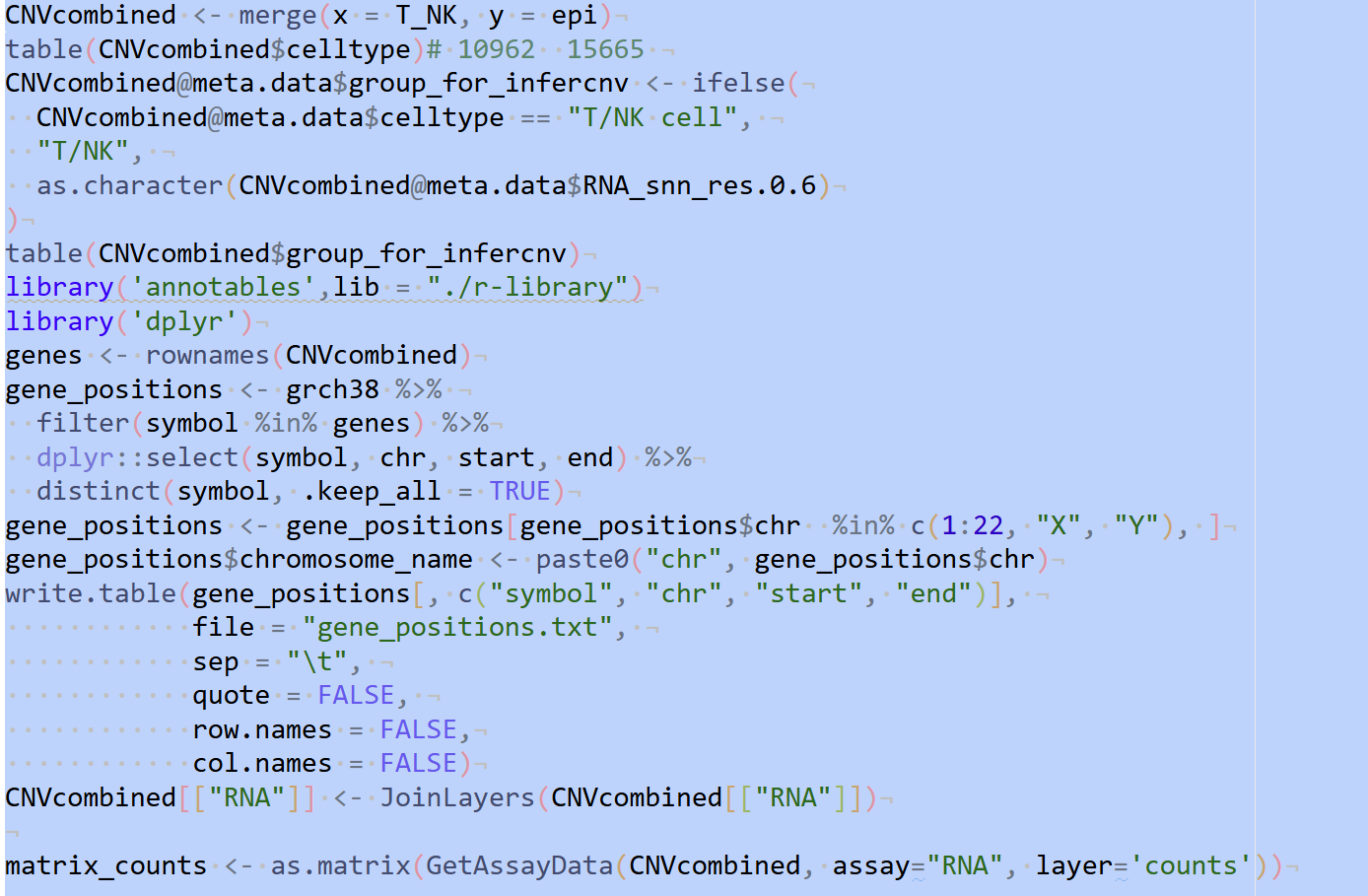
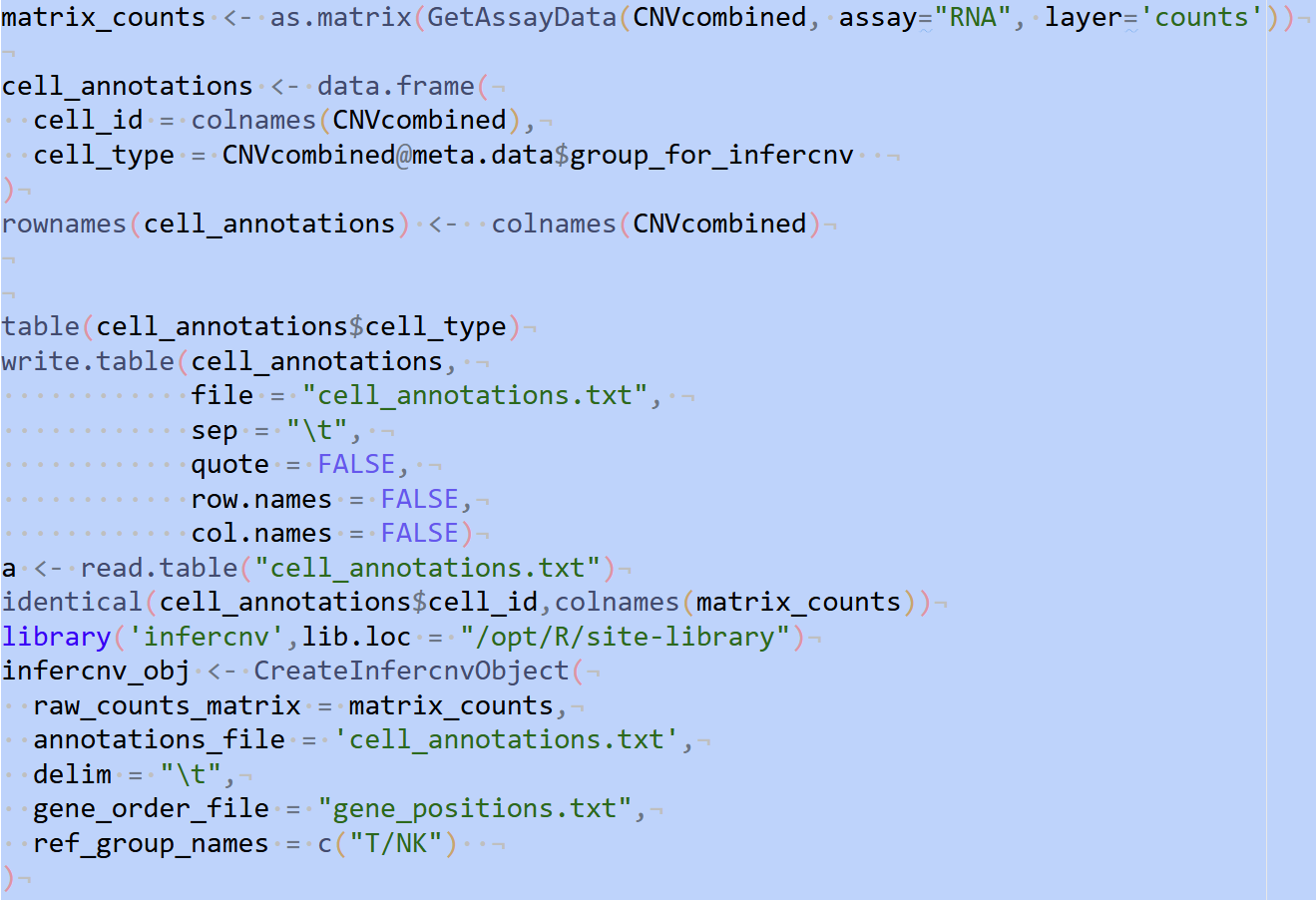
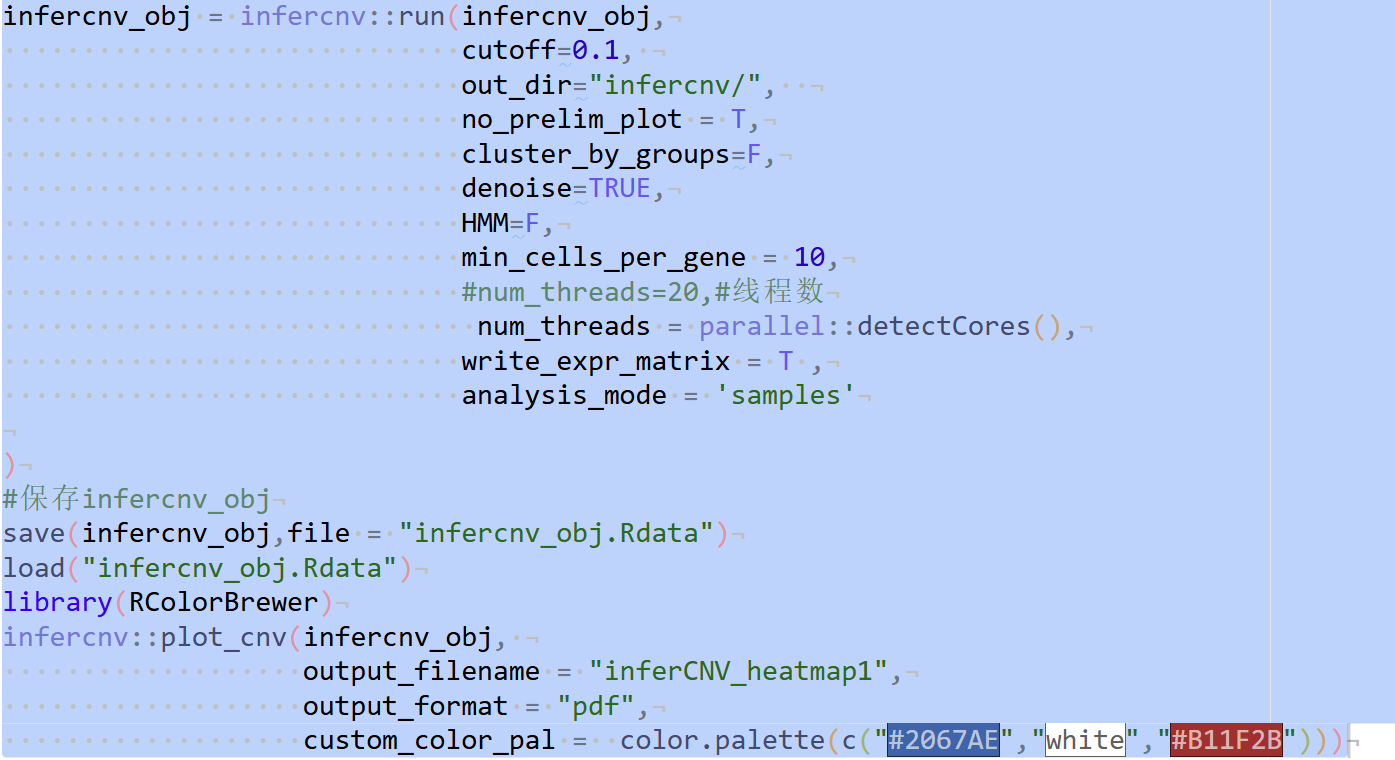
为什么我的inferCNV跑出来分层聚类树里永远只有observed这一种,我的annotation里面明明是T/NK作为参照以及0-9群上皮细胞,想判断0-9群的恶性成都,但是做出来下面observed里面没有0-9群的分群显示




上午好☀️☀️☀️️
本答案参考通义千问
你的问题描述表明你在使用 inferCNV 进行单细胞数据的拷贝数变异(Copy Number Variation, CNV)分析时,遇到了聚类树(dendrogram)中只有 observed 一种标签,而你期望看到的是基于 T/NK 作为参照、0-9群上皮细胞的分层聚类结果。这可能涉及到几个关键点:
inferCNV 的工作原理
inferCNV 主要通过比较不同细胞之间的基因表达模式来推断拷贝数变异。它通常会将细胞分为两类:参考细胞(reference cells) 和 目标细胞(target cells)。
observed 标签的意义
在 inferCNV 的输出中,observed 表示的是 根据拷贝数变异推断出的细胞类型或状态,而不是你原始的注释信息(如 T/NK 或 0-9 群)。如果你没有正确设置参考细胞,或者未在 inferCNV 中指定注释信息,那么 observed 可能无法显示你预期的分类。
为什么没有 0-9 群的分群?
0-9 群的细胞标记为参考细胞(reference cells)。0-9 群被误认为是参考细胞,则不会参与分析。0-9 群的恶性程度,应该将它们设为 目标细胞(target cells),并让 T/NK 作为参考细胞。以下是你需要做的步骤,以确保 inferCNV 正确识别和分析 0-9 群的细胞,并在聚类树中显示正确的分层结构。
确保你的 Seurat 对象中已经正确设置了 ident 或 cluster 注释,例如:
Idents(seu) <- "cell_type" # 假设你有 cell_type 列
注意: inferCNV 不直接使用
ident,而是通过reference_cells参数指定哪些细胞作为参考。
你需要明确告诉 inferCNV 哪些细胞是参考细胞(比如 T/NK),其余为目标细胞(比如 0-9 群)。
# 假设 T/NK 是参考细胞,0-9 是目标细胞
reference_cells <- which(seu$cell_type %in% c("T", "NK"))
target_cells <- which(seu$cell_type %in% paste0(0:9))
library(infercnv)
# 运行 inferCNV
infercnv_obj <- infercnv::infercnv_run(
seu,
reference_cells = reference_cells,
target_cells = target_cells,
n_cores = 4,
verbose = TRUE
)
重要提示:
infercnv_run()会自动计算每个细胞的拷贝数变异,并将其保存在infercnv_obj中。
运行完 inferCNV 后,你可以查看聚类树,此时应该能看到 observed 分类,但这是 inferCNV 推断的拷贝数变异结果,而不是你原始的 0-9 群。
infercnv::plot_dendrogram(infercnv_obj)
注意: 如果你希望将
0-9群与observed关联起来,可以手动将observed结果映射到0-9群。
你可以将 inferCNV 推断的 observed 结果合并到 Seurat 对象中,并用原始注释(如 0-9 群)进行标注:
seu$observed_cnv <- infercnv_obj$predicted_state
seu$group <- ifelse(seu$observed_cnv == "normal", "Normal", "Abnormal")
这样你就可以根据
observed_cnv来判断0-9群的恶性程度。
你可以使用 Seurat 的 DimPlot 或 FeaturePlot 来查看 observed_cnv 与 0-9 群的关系:
DimPlot(seu, group.by = "observed_cnv", split.by = "cell_type")
这个图可以帮助你判断哪些
0-9群表现出异常的拷贝数变异。
| 步骤 | 操作 | 说明 |
|------|------|------|
| 1 | 检查注释 | 确保 cell_type 或 cluster 正确 |
| 2 | 设置参考细胞 | 将 T/NK 设为参考细胞 |
| 3 | 运行 inferCNV | 使用 infercnv_run() 并传入参考/目标细胞 |
| 4 | 查看 dendrogram | 确认 observed 是否包含 0-9 群 |
| 5 | 关联结果 | 将 observed 与 0-9 群结合 |
| 6 | 可视化 | 使用 DimPlot 或 FeaturePlot 展示结果 |
错误1:没有正确设置 reference_cells
reference_cells 包含你定义的参考细胞(如 T/NK)错误2:0-9 群被误认为参考细胞
target_cells 包含你想要分析的 0-9 群错误3:没有将 observed 映射回 0-9 群
seu$observed_cnv 与 0-9 群进行关联分析如果你愿意提供一部分数据或代码片段,我可以进一步帮助你调试具体的问题。