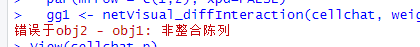
将两个cellchat merge后绘制不同细胞群之间的相互作用数量或相互作用强度报错
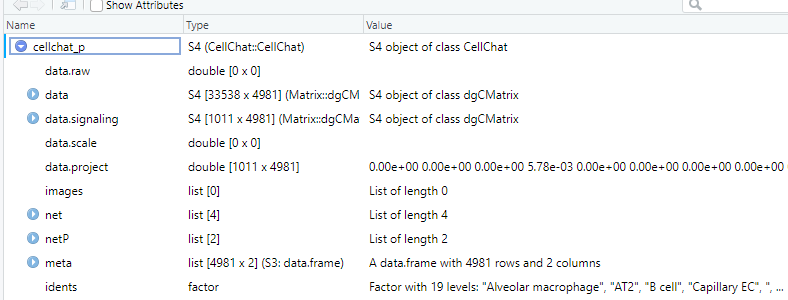
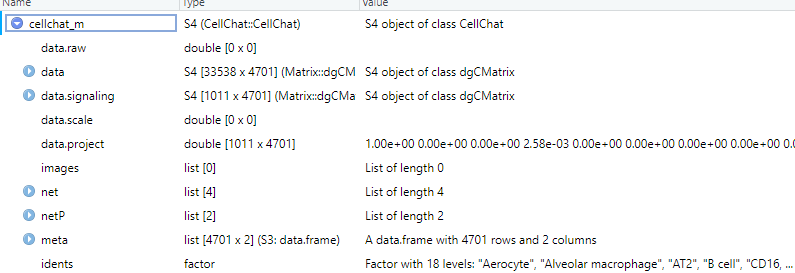
两个cellchat细胞分类不一样

cellchat_p <- readRDS("E:\\R\\RDS3\\Primary_Cellchat.rds")
cellchat_m <- readRDS("E:\\R\\RDS3\\Metastatic_Cellchat.rds")
object.list <- list(cellchat_p, cellchat_m)
cellchat <- mergeCellChat(object.list, add.names = c("Primary", "Metastatic"))
#不同细胞群之间的相互作用数量或相互作用强度不同
pdf(file ="E:\\R\\RDS3\\netVisual_diffInteraction.pdf", width = 25, height =14)
par(mfrow = c(1,2), xpd=FALSE)
gg1 <- netVisual_diffInteraction(cellchat, weight.scale = T)
gg2 <- netVisual_diffInteraction(cellchat, weight.scale = T, measure = "weight")
gg1 + gg2
dev.off()
报错结果: