cnetplot函数可视化富集分析
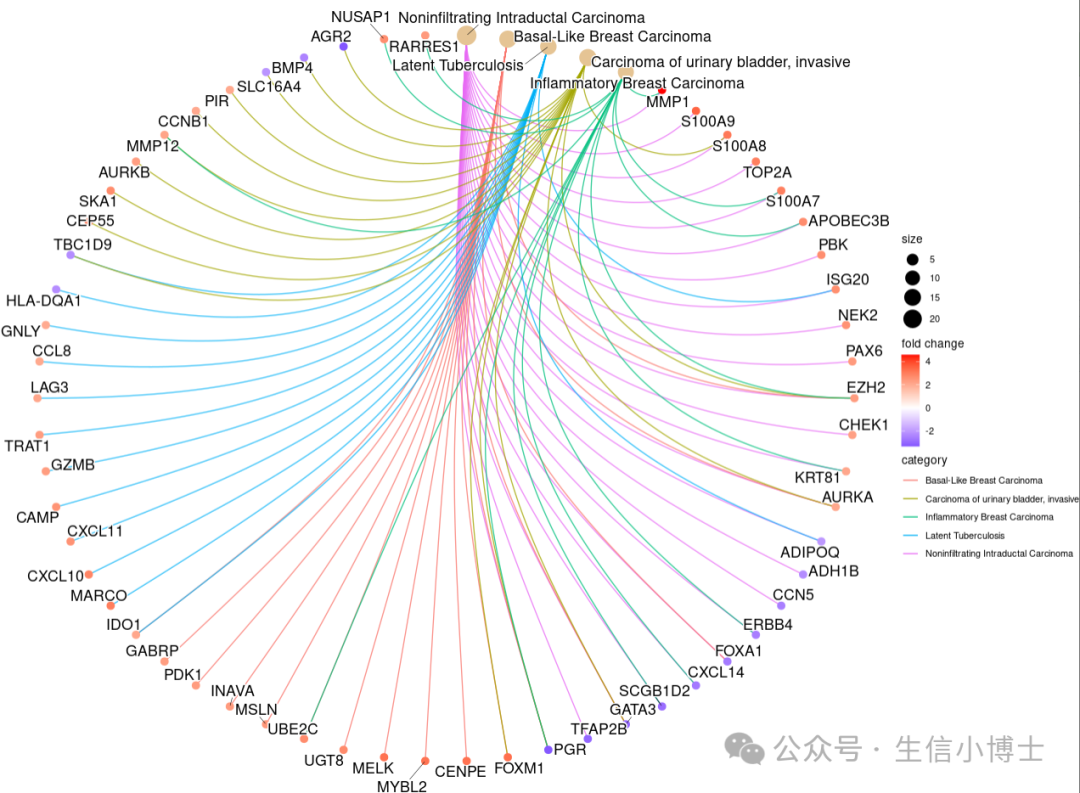
cnetplot(edox, foldChange=geneList, circular = TRUE, colorEdge = TRUE)可视化不出来如下图片,如何调整参数呢
cnetplot函数可视化富集分析
cnetplot(edox, foldChange=geneList, circular = TRUE, colorEdge = TRUE)可视化不出来如下图片,如何调整参数呢

 关注
关注让 “紫雾凌寒” 来帮你回答,本回答结合 DeepSeek
如果还有问题,请评论或点击头像私信,如果有帮助到您, 请关注、采纳、点赞
cnetplot函数无法得到预期的可视化图片。可能原因包括输入数据格式不正确、参数设置不合理等。例如edox数据可能不符合要求的格式,geneList的格式或内容有误,或者circular和colorEdge等参数的设置与数据不匹配。edox数据是正确的富集分析结果对象。它应该包含基因集和相关的富集信息等。例如,如果是基于clusterProfiler包进行富集分析得到的结果,edox应该是enrichResult对象。geneList通常是一个数值向量,其中基因名作为名称,对应的值可以是表达量变化等信息。检查其格式是否正确,基因名是否与edox中的基因一致。circular = TRUE表示绘制圆形的网络,如果数据不适合这种布局可能会导致问题。尝试设置circular = FALSE看看是否能正常绘制普通的网络布局。colorEdge = TRUE用于给边上色,检查是否因为边的相关信息缺失或格式不对导致上色失败。R
library(clusterProfiler)
library(org.Hs.eg.db)
# 假设geneList是基因表达量变化的向量
geneList <- foldChange
# 示例富集分析结果(这里假设已经正确计算得到enrichResult对象edox)
edox <- enrichGO(gene = names(geneList), OrgDb = org.Hs.eg.db, ont = "BP", pAdjustMethod = "BH", qvalueCutoff = 0.05)
# 绘制cnetplot
cnetplot(edox, foldChange = geneList, circular = FALSE, colorEdge = TRUE)cnetplot函数的输入数据edox和geneList的格式是否正确,确保它们符合要求。然后通过调整circular和colorEdge等参数来尝试解决可视化问题。如果还是不行,可能需要进一步检查富集分析的过程是否正确,以及数据是否完整准确。通过逐步排查数据格式和参数设置等方面,有望解决cnetplot函数可视化不出预期图片的问题。 希望以上解答对您有所帮助。如果您有任何疑问,欢迎在评论区提出。