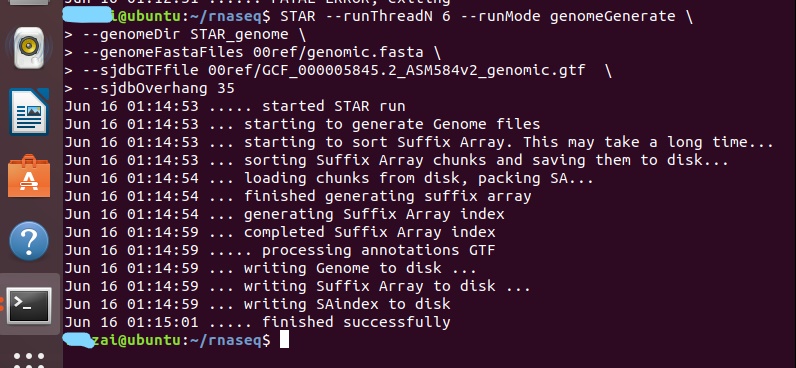
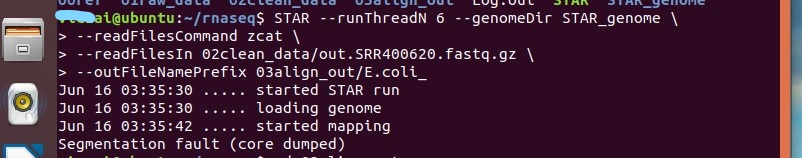
在用STAR做rnaseq序列比对时,出现报错Segmentation fault (core dumped),请各位 解答,谢谢
ai@ubuntu:~/rnaseq$ STAR --runThreadN 6 --genomeDir STAR_genome \
> --readFilesCommand zcat \
> --readFilesIn 02clean_data/out.SRR400620.fastq.gz \
> --outFileNamePrefix 03align_out/E.coli_
Jun 16 03:35:30 ..... started STAR run
Jun 16 03:35:30 ..... loading genome
Jun 16 03:35:42 ..... started mapping
Segmentation fault (core dumped)