GEO下载数据,在ID转换步骤卡死,根据正常方法:
1、有对应R包直接用R包——没有,放弃该法
2、直接下载GPL soft文件用R提取symbol等信息:
出现问题如下
gpl <- getGEO('GPL24676', destdir=".")
colnames(Table(gpl))
gpl <- getGEO('GPL24676', destdir=".")
Using locally cached version of GPL24676 found here:
./GPL24676.soft.gz
colnames(Table(gpl))
character(0)
如图所示,正常GPL应该会出现:
[1] "ID" "SPOT_ID" "CONTROL_TYPE"
[4] "REFSEQ" "GB_ACC" "GENE"
[7] "GENE_SYMBOL" "GENE_NAME" "UNIGENE_ID"
[10] "ENSEMBL_ID" "TIGR_ID" "ACCESSION_STRING"
[13] "CHROMOSOMAL_LOCATION" "CYTOBAND" "DESCRIPTION"
[16] "GO_ID" "SEQUENCE"
此类信息
但GPL24676运行出来col为0,直接卡死
3、搜芯片来源,发现其为Illumina NovaSeq 6000 (Homo sapiens),0相关贴,卡死
4、尝试过这种注释信息不全的万能法(如下图)了,也失败了,卡在提取下载的GPL数据的步骤上,它是还是0
BiocManager::install("clusterProfiler")
BiocManager::install("org.Hs.eg.db")
求问,这种数据怎么进行基因ID转换!
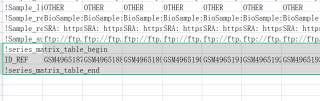
上图是GSE的矩阵,可以看到和别的数据不一样,它的start和end中间没有任何数据。
SOS!

