我是用 CHO2017 公开数据集进行左、右手运动想象任务的溯源,然后使用谢菲尔德2018分割文件对脑区进行分割并使用 pca_flip 提取时间特征。最后放入FBCNet、FBCSP中进行分类,可是结果与EEG信号进行分类 分别相差大概10个点、6个点。是哪里出了问题呢?我该学习哪方面的知识,请赐教!
以下是溯源部分的代码
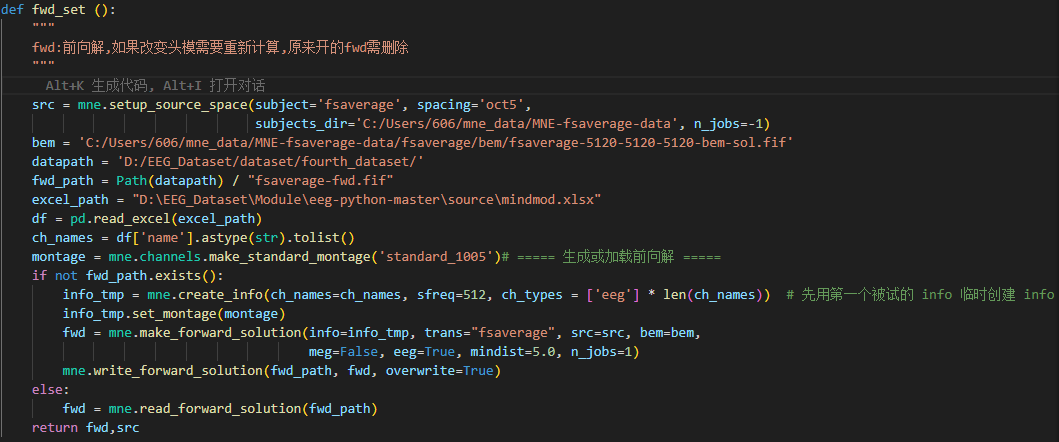
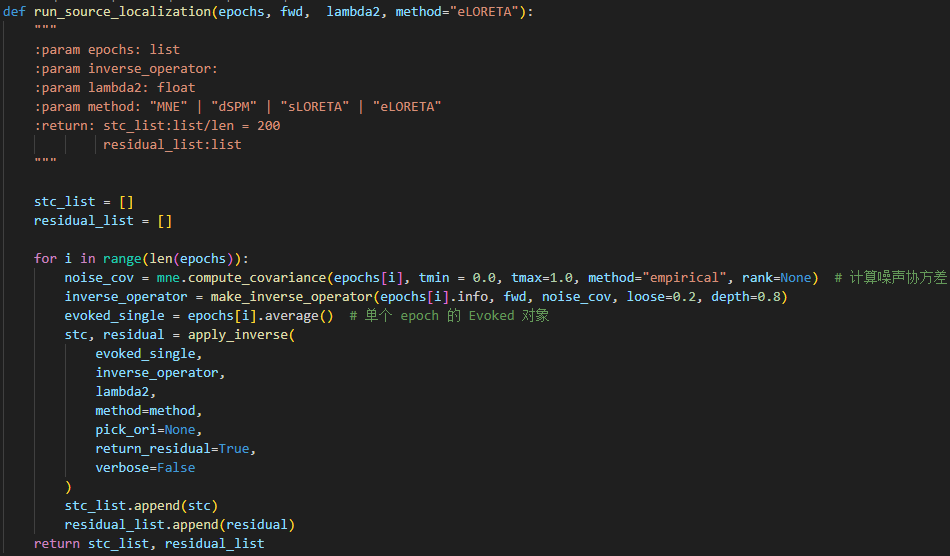
我感觉可能是溯源上出了问题
我是用 CHO2017 公开数据集进行左、右手运动想象任务的溯源,然后使用谢菲尔德2018分割文件对脑区进行分割并使用 pca_flip 提取时间特征。最后放入FBCNet、FBCSP中进行分类,可是结果与EEG信号进行分类 分别相差大概10个点、6个点。是哪里出了问题呢?我该学习哪方面的知识,请赐教!
以下是溯源部分的代码


我感觉可能是溯源上出了问题
晚上好🌙🌙🌙
本答案参考通义千问
你目前的实验流程涉及运动想象(Motor Imagery, MI)源定位、脑区分割、特征提取与分类等多个环节,这些步骤中任何一个环节都可能影响最终的分类准确率。根据你的描述,使用 FBCNet 和 FBCSP 进行分类时,与直接使用 EEG 信号进行分类相比,准确率分别低了约 10% 和 6%,这说明在特征提取或源定位阶段可能存在一些问题。
mne.source_estimate)。重点:源定位质量直接影响特征提取效果。
重点:ROI 的选择应基于任务相关脑区(如运动皮层)并结合先验知识。
重点:确保特征提取过程符合任务需求,并且与分类器匹配。
重点:分类器的输入特征必须经过适当预处理。
为了提升你的研究水平,建议你学习以下方面的知识:
import mne
# 加载源估计数据
src = mne.read_source_spaces('subject-1-src.fif')
# 绘制源空间
mne.viz.plot_3d_source_space(src, subject='subject-1', subjects_dir=subjects_dir)
aparc+aseg.mgz)进行 ROI 提取。from nilearn import datasets
from nilearn import image
# 加载脑区分割图
atlas = datasets.fetch_atlas_schaefer_2018(resolution=200)
labels = image.load_img(atlas['maps'])
[channel, time])。from sklearn.decomposition import PCA
from sklearn.preprocessing import StandardScaler
# 假设 X 是 [trial, channel, time] 的数据
X = X.reshape(X.shape[0], -1) # 展平为 [trial, channel*time]
scaler = StandardScaler()
X_scaled = scaler.fit_transform(X)
pca = PCA(n_components=50)
X_pca = pca.fit_transform(X_scaled)
from sklearn.model_selection import cross_val_score
from sklearn.svm import SVC
model = SVC(kernel='linear')
scores = cross_val_score(model, X_pca, y, cv=5)
print("Accuracy: %0.2f (+/- %0.2f)" % (scores.mean(), scores.std() * 2))
| 问题点 | 原因 | 解决方案 | |--------|------|----------| | 源定位质量差 | 头模型不准确或正则化参数不当 | 使用 MNE 或 FieldTrip 进行源定位,验证结果 | | 脑区分割不合理 | ROI 选择不当 | 使用 FreeSurfer 或 FSL 提取感兴趣区域 | | 特征提取不佳 | PCA_flip 参数设置错误 | 优化特征提取流程,尝试 CSP 或 FBCSP | | 分类器配置不当 | 特征未标准化或训练策略错误 | 标准化特征,使用交叉验证评估模型 |
如果你愿意分享具体的代码片段或数据结构,我可以进一步帮你分析具体哪一步出现了偏差。希望以上内容对你有所帮助!